O navegador do genoma do UCSC faz 25 anos
Jim Kent sabia uma coisa ou duas sobre codificação. Veterano dos primeiros dias da computação pessoal, Kent desenvolveu software para computadores Amiga, Atari e IBM ao longo dos anos 80, incluindo ferramentas pioneiras para animação por computador.
Em 2000, Kent havia dado o salto para a bioinformática, desenvolvendo o sistema de navegação de genoma computacional que se tornaria conhecido como navegador genoma da Universidade da Califórnia em Santa Cruz (UCSC). Mas suas expectativas foram tingidas por sua experiência no setor. “Eu estava realmente acostumado a software com duração de cerca de três anos”, diz Kent. Sua última criação, ele previu, não se sairia de maneira diferente.
Toda base em todos os lugares de uma só vez: a pangenômica chega à maioridade
“Isso não aconteceu”, diz o geneticista David Haussler, diretor científico do UCSC Genomics Institute que administra o navegador do genoma. Este mês marca o 25º aniversário do navegador, que floresceu em um recurso essencial para biólogos em todo o mundo. Em qualquer dia, mais de 7.000 usuários únicos se envolvem com o site, que hospeda dados do genoma de humanos, ratos e mais de 100 outras espécies e vírus animais, e permite que os pesquisadores anotem e interrogem essas seqüências de inúmeras maneiras.
O genomista comparativo Michael Hiller, na Goethe University, em Frankfurt, na Alemanha, conta com o navegador para investigar padrões de mudança de genoma entre espécies animais para identificar assinaturas de evolução. “Olhar para o navegador do genoma é uma grande parte do que nós e os outros membros do laboratório fazemos diariamente”, diz ele. Ele até incorporou a ferramenta em um curso que ensina. “Isso faz parte dos exercícios”, diz ele.
Com um fluxo constante de atualizações e ferramentas de anotação gerada pelo usuário, o navegador do genoma não mostra sinais de desaceleração. “O site ganhou vida própria”, diz Max Haeussler, o principal investigador do projeto. Mas, como em muitos projetos científicos dos EUA em 2025, a ameaça real à sua longevidade vem do aperto das cordas da bolsa, com cortes maciços e reestruturação nos Institutos Nacionais de Saúde dos EUA (NIH) colocando esse recurso essencial em risco.
Atingindo o chão correndo
Tudo começou com o projeto do genoma humano. Haussler ingressou no esforço no final de 1999, com o objetivo de desenvolver software que pudesse identificar e anotar genes no genoma recém -montado. Mas havia um problema. “Ficou claro para mim muito rapidamente que esse problema de montar o genoma humano a partir de pequenos pedaços de DNA sequenciado era extremamente difícil, e eles não tinham um plano sério para fazê -lo”, lembra Haussler. Ele se virou para Kent, um codificador qualificado que estava concluindo um doutorado na UCSC, focado no desenvolvimento do Intronerator, um navegador de genoma para mapear a expressão de genes na espécie do modelo de worm Caenorhabditis elegans.
Em uma trituração frenética de programação que durou várias semanas em 2000, Kent desenvolveu Gigassembler – 10.000 linhas de código que Haussler descreve como “o assembler que salvou o dia”. Permitiu que o Projeto Genoma Humano concluísse seu primeiro rascunho a tempo de sua apresentação oficial em 26 de junho de 2000.

Jim Kent bioinformático, que ajudou a desenvolver o navegador do genoma do UCSC.Crédito: Don Harris/UC Santa Cruz Serviços de foto
O interesse inicial de Kent era principalmente pragmático. “Eu queria levantar o navegador do genoma humano apenas para poder verificar novamente minha assembléia”, diz ele. Mas quando ele e Haussler lançaram a primeira iteração do navegador do genoma da UCSC em 7 de julho de 2000, foi um sucesso instantâneo. A comunidade científica baixou coletivamente aproximadamente metade de um terabyte de dados nas primeiras 24 horas da operação do navegador, estima a equipe – uma quantidade impressionante na época.
O genoma estava então apenas cerca de 90% completo e repleto de lacunas, mas o mundo finalmente tinha uma janela disponível gratuitamente para examiná -la. “Apenas ser capaz de ir a uma visão do navegador das primeiras anotações de genes ou da clonagem precoce era como um ímã”, diz Barbara Wold, bióloga molecular do Instituto de Tecnologia da Califórnia em Pasadena. “Isso atraiu todo mundo para a comunidade de usuários.” É importante ressaltar que o navegador também estabeleceu ‘marcadores de milha’ que os pesquisadores poderiam usar para mapear descobertas e variantes em genomas individuais.
Ian Holmes, biólogo computacional da Universidade da Califórnia, Berkeley, também credita Kent e a equipe da UCSC por normalizar a idéia de que dados genômicos devem ser livres e fáceis de se envolver e reutilizar. “A ideia de que tudo deve estar disponível na web e não os aplicativos de desktop ainda era uma idéia extremamente radical”, diz Holmes. “Eles inventaram uma tonelada de formatos de arquivo e apenas maneiras de armazenar dados que estamos usando amplamente hoje.”

O senador Tom Harkin (Democrata) ajudou a garantir financiamento para o projeto do genoma humano, capaz de montar a sequência usando o código da equipe do navegador do genoma do UCSC.Crédito: NHGRI/NIH
Nos anos seguintes, o navegador do genoma atualizou seus mapas com os dados mais recentes do projeto do genoma humano, bem como de outras espécies modelo populares, incluindo o mouse e o rato. Kent também introduziu um recurso definidor, com base em sua experiência com o Intronerator: a capacidade de os usuários gerarem e compartilharem ‘faixas’ personalizadas que anotam seletivamente os principais recursos do genoma. As faixas são mapas interativos de estradas visuais, apresentados ao lado do genoma, que traçam a localização de características importantes, como elementos regulatórios e variantes de genes associados à doença. “Estávamos muito à frente de todos com esse recurso, e acho que foi por isso que acabamos no meio do ecossistema”, diz Kent.
O esforço ganhou impulso precoce graças à adesão de consórcios internacionais importantes, como o projeto Enciclopédia dos Elementos de DNA (Encode). A codificação foi focada nos motivos funcionais que povoam os 98,5% do genoma humano que não contém sequências genéticas codificadoras de proteínas. Haussler diz que a lista de faixas personalizadas do navegador do genoma “cresceu dramaticamente” durante o trabalho da equipe da codificação, permitindo que os usuários visualizem os muitos promotores, intensificadores e RNAs que não codificam proteínas que o consórcio descobriu.
Um recurso durável
Ainda assim, a previsão sombria de Kent da expectativa de vida do navegador não era irracional – essas narrativas são típicas na academia. “Imagine se o Microsoft Office desaparecer a cada três anos”, diz Haeussler. “Este é o caso da maioria dos pacotes de software de bioinformática.” Os estudantes de pós -graduação e os pesquisadores de pós -doutorado seguem em frente, e as plataformas estagnam mesmo à medida que novos dados se acumulam, até que alguém seja obrigado a desenvolver uma substituição.
Como o genoma humano transformou o estudo de doenças raras
E os dados investiram constantemente. “Não há mais reuniões da Casa Branca quando você publica um genoma”, diz Haeussler, referindo -se ao anúncio do primeiro rascunho do genoma humano. “Agora recebemos um por dia ou até dez por dia.” E não são apenas genomas, mas também novas camadas de informação que as pessoas desejam mapear em cima delas, como transcrições de RNA completas e sinais epigenéticos que desempenham um papel central na regulação de genes. Ao mesmo tempo, a base de usuários da ferramenta cresceu constantemente.
A capacidade do navegador do genoma do UCSC de sobreviver e acomodar esse crescimento é parcialmente atribuível à decisão inicial de construir uma equipe de programadores e engenheiros dedicados. Muitos deles foram veteranos experientes no Vale do Silício impressionados com as ambições do navegador e a história de origem robusta. “Eu meio que caí na defesa de doutorado (de Kent)”, diz Angie Hinrichs, uma ex -designer de microchip que tinha experiência mínima de biologia, mas, no entanto, se ofereceu para a equipe de navegador do genoma no início de 2002. Ela seria uma das primeiras novas contratações de uma equipe que inchou aproximadamente 30 pessoas no auge.
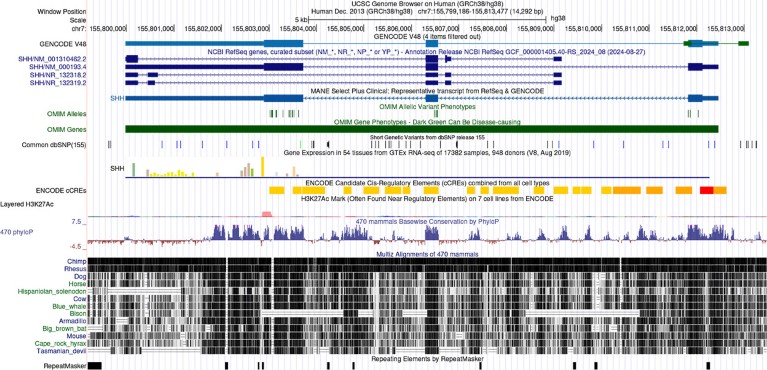
Saída do navegador do genoma do UCSC mostrando informações para o humano Shh Gene, que está envolvido no desenvolvimento embrionário.Crédito: Perez et al. O banco de dados do navegador do genoma do UCSC: atualização 2025. Pesquisa de ácidos nucleicos 2025 PMID
O acesso a infraestrutura de computação acessível e poderosa também não dói. “Nos primeiros dias, provavelmente parecia muito ter 32 processadores, e agora nossa máquina de desenvolvimento tem 192”, diz Hinrichs. Os dados em si, assim como o código para executar o navegador, residem em matrizes em escala de petabytes que incluem inúmeros discos rígidos de estado sólido e super velocidade, acrescenta ela.
A base de código do navegador do genoma cresceu de apenas 10.000 linhas para mais de três milhões, diz Haussler. Mas sua arquitetura fundamental não mudou muito em 25 anos. Hinrichs diz: “Houve muitos chefs na cozinha, e ficou um pouco bagunçado, mas a estrutura central dela era tão limpa que realmente funcionou e ainda podemos nos desenvolver. Credito Jim Kent por isso”.
Do ponto de vista de Kent, essa durabilidade é atribuída ao seu foco na criação de código modular que pode ser ajustado sem derrubar todo o edifício, além de um corpo de testes dedicado. “Tínhamos uma equipe de garantia de qualidade que era cerca da metade do tamanho da equipe de desenvolvimento de software”, diz ele. “Acho que só tivemos que reverter uma versão anterior uma vez.”
Projeto Genoma Humano-O editor-chefe da Nature reflete 20 anos em
A equipe teve que criar maneiras mais eficientes de lidar com dados, no entanto. Em 2011, por exemplo, introduziu o formato ‘hub de track’ como uma solução para as faixas personalizadas cada vez mais grandes e complexas que os laboratórios externos estavam desenvolvendo para a anotação do genoma. Os hubs da faixa compreendem conjuntos de arquivos binários relativamente compactos que são hospedados pelos próprios criadores de dados, e não na UCSC. Os dados são transmitidos sob demanda e “os formatos de arquivo de Jim são projetados para que você possa pular entre eles”, diz Haeussler. A capacidade de acessar rápida e seletivamente as partes relevantes dos dados hospedados remotamente minimiza a quantidade que precisa fluir entre o usuário e o navegador do genoma. Isso mantém a velocidade de desempenho, mesmo quando o número de faixas aumenta. “Temos milhares de laboratórios fazendo -os, mas a maioria deles nunca vemos”, diz Haeussler.
E em 2023, a equipe lançou a iniciativa Genark (Genome Archive) para acomodar o influxo interminável de novas sequências de genoma (H. Clawson et al. Genoma Biol. 24217; 2023). Esse sistema o torna simples para o navegador genoma retirar os genomas recentemente enviados do banco de dados de montagem hospedado pelo Centro Nacional de Informações de Biotecnologia dos EUA (NCBI), permitindo que os pesquisadores visualizem e anotem esses genomas. Atualmente, a Genark hospeda mais de 6.000 genomas, variando de bactérias a primatas.




