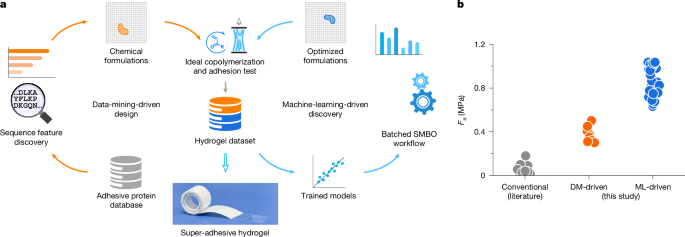
Projeto de novo orientado a dados de hidrogéis super-adesivos
Fabricação de hidrogel
Todos os géis de copolímeros foram sintetizados por copolimerização de monômeros de monômeros de um etapa de um etapa de monômeros com um reticulador químico. A concentração de reticulação foi fixada a 0,1 mol% em relação ao conteúdo total de monômero para equilibrar a elasticidade e a deformabilidade dos géis27. Soluções de DMSO contendo monômeros funcionais (concentração total de 2,4 m) com composições derivadas de DM e ML (Tabelas Suplementares 2 e 7), Linker químico (glicerol 1,3-diacrylate, 2,4 mm) e UV iniciador (2-oxoglutrásico, 6 mm) foram usados. Por exemplo, para preparar o gel G-Max, 1,819 g de Ba, 0,413 g de HOLE, 0,264 g de CBEA, 0,561 g de ATAC, 0,441 g de ervilha, 8,4 mg de glicerol 1,3-dicrylate a 10 mg de 2-oxmão, digilaterol a 2,8 mg de voletol, digilaterol Diacrylate a 10 mg de voletol a ácidos de 2,8 mg de voletol, foram adicionados a digrylato de 2,8 mg de voletol, foram adicionados a digilato de 2,8 mg de voletol, foram adicionados a um digiltetrol de 2,8 mg de voletol, foram adicionados a um digiltetrol de 2,8 mg de voletol, foram adicionados a um digiltetrol de 2,8 mg de 2-oxogltetral a acidão de diglutol. A solução precursora foi transferida para uma caixa de luvas para remover oxigênio, derramada em uma célula de reação (duas placas de vidro de 10 cm × 10 cm, espaçamento de 0,5 mm) e irradiado com luz UV (comprimento de onda de 365 nm, 4 MW CM CM−2 intensidade) para 8 h para formar géis (Fig. 9A suplementar). Após a irradiação UV, mais de 99% dos monômeros foram convertidos em polímeros, conforme confirmado por RMN (Fig. 9B suplementar).
Os organogéis preparados foram então imersos em solução salina normal (NaCl de 0,154 M) para remover solventes e produtos químicos residuais, com a solução salina trocada a cada 12 h por pelo menos 2 semanas até que o equilíbrio do inchaço fosse atingido. Os hidrogéis foram armazenados em solução salina normal antes do uso.
Caracterização de adesão subaquática
O teste de aderência foi realizado usando um testador Shimadzu (Autograph AG-X) equipado com o software Trapezium X. O hidrogel (0,3-0,8 mm de espessura) no equilíbrio do inchaço foi aderido à sonda usando adesivo de cianoacrilato (super cola). Para triagem rápida, hidrogéis acionados por DM da rodada de treinamento e hidrogéis acionados por ML de três rodadas de otimização foram preparados como amostras de 15 mm de diâmetro. Para estudos detalhados de adesão, amostras de 10 mm de diâmetro foram usadas para evitar exceder a faixa de força do instrumento. Essa mudança de diâmetro não afetou os resultados da força do adesivo. O hidrogel na sonda foi então imerso em uma solução de teste (por exemplo, solução salina normal) por 5 min para atingir o equilíbrio. A sonda desceu em direção ao substrato a 1 mm min-1 Até que uma força de carregamento de 10 N fosse aplicada, mantida por 10 s e retirada a 10 mm min min-1 (Fig. 10). Essas condições de teste foram usadas como um protocolo padrão, a menos que especificado de outra forma. Para testes de adesão repetidos, os hidrogéis descansaram debaixo d’água por 5 minutos entre os ciclos, com substratos de vidro substituídos a cada 100 testes. Para ciclos prolongados de fixação -detenção (dados estendidos Fig. 8), uma força de carregamento de 5 n e um tempo de contato de 10 s foram usados para minimizar a fadiga do gel. Cada amostra foi testada pelo menos três vezes. Para a construção do conjunto de dados de hidrogel, a maior resistência adesiva registrada para cada amostra foi relatada como Fumrepresentando o desempenho máximo de adesão sob as condições específicas.
A força adesiva de cisalhamento da volta foi medida usando uma máquina de teste universal (UTM, Instron 5965). Um hidrogel (10 mm de diâmetro, área UM= 78,5 mm2) no inchaço, o equilíbrio foi imprensado entre duas lâminas de vidro, pressionadas a 20 N por 1 min em solução salina normal. O carregamento de cisalhamento foi aplicado a 50 mm min-1. Força adesiva de cisalhamento (Fum) foi calculado como Fum=Fmáx / UMondeFmáxé a força de carga máxima. Para testes de durabilidade da adesão (Fig. 15 suplementar), o conjunto sanduíche foi armazenado em solução salina normal para durações variadas antes do teste.
A resistência interfacial foi medida em testes de descamação de 180 ° usando o Instron 5965. As tiras de hidrogel (10 mm × 150 mm) foram aderidas a um substrato de vidro em solução salina normal usando pressão leve, seguida por um rolo de mão de 2 kg aplicado em cada direção por 1 min para garantir um contato uniforme. Os filmes de polietileno tereftalato (PET) (espessura de 50 μm) serviram como um apoio rígido. Os testes de descamação foram realizados a 50 mm min-1. Resistência interfacial ( Gc) foi calculado comoGc = 2Fc/ c onde Fcé a força do platô e cé a largura da amostra (10 mm).
DM de proteínas adesivas
Um conjunto de dados abrangente das proteínas adesivas foi compilado a partir do banco de dados de proteínas NCBI, usando ‘proteínas adesivas’ como palavra -chave consulta. Um total de 24.707 seqüências de proteínas de 3.822 organismos diferentes (bactérias, vírus, eucariotos e animais) foram coletados sem limpeza adicional de dados. Com base nas anotações de taxonomia, as proteínas foram agrupadas por espécies e uma sequência de consenso foi gerada para cada espécie capturar padrões de sequência comum e reduzir a influência de variações individuais.
O conjunto de dados incluiu 3.111 espécies, observando que a sobreposição taxonômica resulta em contagens de proteínas que não somam 24.707. Para análises robustas, as 200 principais espécies, classificadas pelo número de proteínas distintas identificadas por espécie, foram selecionadas para estudos posteriores.
As sequências de proteínas foram exportadas em formato de fasta45 Usando a interface bio.seqio em biopython46. Sequências de consenso foram calculadas com ômega clustal23que executa o alinhamento de múltiplas seqüências gerando uma matriz de distância a partir de alinhamentos em pares, construindo uma árvore -guia com base em relações evolutivas e alinhando progressivamente sequências das mais próximas às mais distantes. O alinhamento resultante identifica os resíduos mais frequentes em cada posição, produzindo uma sequência de consenso que destaca regiões conservadas.
CLUSTAL OMEGA foi executado com o comando:
$$./{\ rm {c}} {\ rm {l}} {\ rm {u}} {\ rm {s}} {\ rm {t}} {\ rm {a}} {} {}} {} {{\ ge \ mbox {-} {\ rm {i}} \, {\ rm {\ mbox {“} }} {\ rm {i}} {\ rm {n}} {\ rm {p}} {\ rm {u}} {\ rm {t}} {\ rm {\ _} am \ mbox {-} \ mbox {-} {\ rm {o}} {\ rm {u}} {\ rm {t}} {\ rm {f}} {\ rm {m} } {\ rm {t}} \, = \, {\ rm {c}} {\ rm {l}} {\ rm {u}} \, \ mbox {-} {\ rm {o}} \, {\ rm {\ mbox {“} }} {\ rm {o}} {\ rm {u}} {\ rm {t}} {\ rm {p}} { \ rm {u}} {\ rm {t}} {\ rm {\ _}} {\ rm {a}} {\ rm {l}}{\rm{n}}{\rm{\_}}{\rm{f}}{\rm{i}}{\rm{l}}{\rm{e}}{\rm{\mbox{”}}}\, \ mbox {-} {\ rm {v}} $$
onde “input_file” e “output_aln_file” denotam as sequências de proteínas de entrada e sequências de consenso de saída, respectivamente. As 200 seqüências de consenso geradas foram utilizadas para análise de sequência subsequente e projeto de formulação de hidrogel.
Métodos ML
Um vetor de característica tridimensional,ϕeu= ( ϕBaAssim, ϕHEAAssim, ϕCBEAAssim, ϕATACAssim, ϕAamAssim, ϕERVILHA), foi usado para representar proporções de monômero em hidrogéis. A variável alvo era força adesiva, Fum . Para modelar a relação entre ϕeu e Fum Exploramos os modelos Linear e não linear ML (tabelas suplementares 5 e 6).
Os modelos lineares incluíram menos regressão absoluta de regressão do operador de seleção e regressão de cume (cume). Modelos não lineares compreendidos k-Ear os vizinhos mais conhecidos (KNN), regressão de Kernel Ridge (KRR), regressão vetorial de suporte (SVR), regressão da floresta aleatória (RFR), regressão de aumento de gradiente com XGBOOST (XGB), regressão de árvores extras (ETR) e processo gaussiano (GP) com um matérn kernnel32,34. Esses modelos não lineares incluem abordagens não paramétricas (KNN), baseadas em kernel (KRR, SVR e GP) e de sequência de árvores (RFR, XGB e ETR), permitindo uma comparação abrangente34,35,47.
O XGB era de V.1.6.2, enquanto os outros modelos foram implementados usando o Scikit-Learn (v.1.0.2) e o Scikit-Otimize (v.0.9.0). O hyperparameter n_estimators foi ajustado usando optuna48enquanto outros foram otimizados usando a pesquisa de grade (Tabela 6 suplementar). Uma estratégia de validação cruzada de 10 vezes foi usada para avaliar o desempenho preditivo em nosso conjunto de dados de 180 hidrogéis, usando o erro quadrado médio da raiz (RMSE) como a métrica. GP e RFR, com o menor RMSE em erro de teste de treinamento usando uma divisão de trem/teste de 90%/10% (dados estendidos Fig. 4), emergiu como o melhor desempenho e o segundo colocado, respectivamente, e foram posteriormente usados como modelos de base (substituto).
Para fazer previsões extrapolativas, tentamos três tipos de métodos.
-
1.
Enumeração somente de exploração:
Dez milhões ϕeu Os vetores foram gerados a partir de uma distribuição uniforme (0, 1,0) para cada monômero, normalizado para soma para 1,0. Os cinco principais vetores, classificados por previstos Fum de cada modelo, foram validados experimentalmente.
-
2.
Bo em lote:
-
GP_KB: usou previsões de GP como valores hipotéticos para selecionar os próximos pontos de dados que maximizam o EI.
-
Gp_clmax: usado o máximo Fum (Y_MAX) do conjunto de treinamento como um valor hipotético para selecionar os próximos pontos de dados com o EI Maximums.
-
Gp_clmin: usado o mínimo Fum (y_min) para selecionar os próximos pontos de dados com o EI Maxima.
-
GP_LP: incorporou um termo penalizado localmente no cálculo da EI37.
Gp_kb, gp_clmax e gp_clmin simplificaram a junta q-EI Cálculo de probabilidade36 Usando o valor de previsão do GP como um valor hipotético para selecionar os próximos pontos de dados com o EI Maxyums. Um tamanho de lote deq= 10 foi selecionado.
-
-
3.
Otimização seqüencial baseada em modelo em lotes (SMBO):
-
GP-RFR: GP como provedor de valor hipotético e RFR como o maximizador EI.
-
RFR-RFR: RFR como provedor de valor hipotético e o Maximizador EI.
-
RFR-GP: RFR como provedor de valor hipotético e GP como o maximizador EI.
-
RFR-GP*: RFR-GP Com uma partida quente, 10 pontos gerados por RFR foram adicionados ao conjunto de dados real para a regressão GP.
-
RFR-ERT: RFR como provedor de valor hipotético e ETR como o maximizador EI.
-
RFR-GBM: RFR como provedor de valor hipotético e GBM como o maximizador EI.
A SMBO atualiza iterativamente o modelo substituto enquanto explora pontos de dados promissores33. GP e RFR, quando usados como provedores de valor hipotético, exploração e exploração de equilíbrio, enquanto GP_Clmax e GP_Clmin enfatizam a exploração e a exploração, respectivamente49.
-
SMBO (algoritmo suplementar 1) consiste em quatro componentes: a verdadeira função (f), domínio global (X), função de aquisição (S) e modelo substituto (M). Dados de treinamento inicial ( D ) são amostrados de X e experimental FumOs valores são obtidos (linha 1). O modelo substituto M está ajustado a D (linha 3) e S (EI) identifica o próximo ponto de dados com base na incerteza preditiva (linha 4). Este ponto de dados é posteriormente validado experimentalmente (linha 5), atualizando D(linha 6) paraTiterações (linha 2).
EI quantifica melhorias esperadas, \ ({\ int} _ {y*}^{\ infty} (y- {y}^{*}) p (y) {\ rm {d}} y \)sobre o melhor alvo atual ( y*). Devido à natureza intensiva da fabricação de hidrogel (cada um leva cerca de 2 semanas), GP e RFR foram usados como provedores de valor hipotético, permitindo a maximização da junta q-EI Probabilidade sem exigir novos experimentos por iteração. Os maximizadores de EI (GP, RFR, ETR e GBM) usaram hiperparâmetros da Scikit-Otimize (v.0.9.0).
Para o GP como o maximizador EI, o algoritmo de Broyden-Fletcher-Goldfarb-Shannon (L-BFGS-B) GoldFarb-Shannon (L-BFGS-B)50 foi executado 20 vezes por iteração (40 iterações no total) para identificar o ponto com o maior EI, atualizando o GP anterior. Para os outros três maximizadores de EI (RFR, ETR e GBM), 10.000 pontos foram amostrados aleatoriamente por iteração, pois a otimização numérica é mais adequada para modelos de serem de árvores sem informações de gradiente. O SMBO correu para 40 iterações com cada maximizador EI, selecionando dois conjuntos de 10 pontos de dados em cada iteração: os 10 principais classificados pelos valores de EI (tamanho do lote q= 10) e os 10 principais classificados por previstoFumValores para validação experimental. Esses dois conjuntos podem se sobrepor e o número total de pontos de dados pode ser menor que 20.
Para métodos BO (GP_KB, GP_CLMAX, GP_CLmin e GP_LP), o procedimento foi semelhante, exceto que o provedor de valor hipotético era o próprio GP (GP_KB e GP_LP) ou valores constantes (Y_Max para GP_Clmax e Y_Min para GP_Clmin).
Após a primeira rodada, 109 pontos validados expandiram o conjunto de dados para 289 hidrogéis. A segunda e a terceira rodadas adicionaram 27 e 25 pontos, respectivamente, resultando em um conjunto de dados final compreendendo 341 hidrogéis.




